模式识别-非参数技术笔记
非参数技术
K 最近邻算法
原理
K 最近邻算法(KNN)的原理很简单,我们考虑我们已知数据

这是我们现有的数据,假如说存在一个神秘的水果
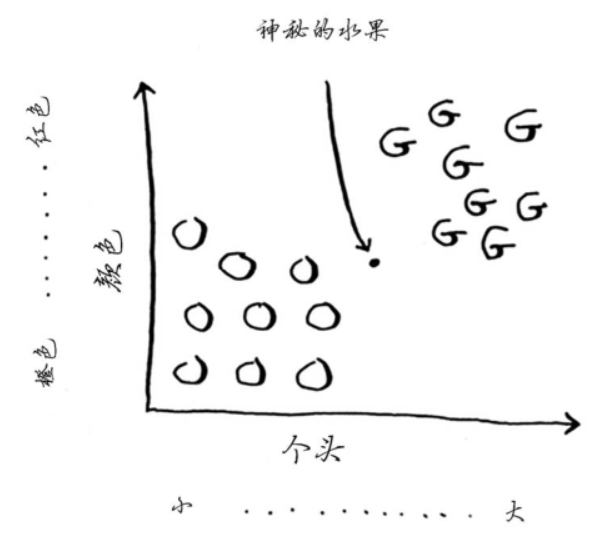
我们判断它是橙子还是柚子的依据就是,来看谁是它最多的邻居。
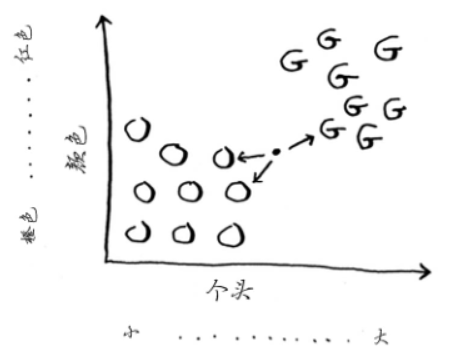
我们可以计算得到,在三个邻居范围内,橙子比柚子多,所以,这个水果很有可能是橙子。
这就是 KNN 算法的基本原理,K 是其中的三。
距离度量
闵可夫斯基距离公式为 \[ L_p(\boldsymbol{x}_i,\boldsymbol{x}_j)=(\sum_{l=1}^n|x_i^{(l)}-x_j^{(l)}|^p)^{\frac{1}{p}} \] 如果 \(p=2\),那么就是欧式距离 \[ L_2(\boldsymbol{x}_i,\boldsymbol{x}_j)=(\sum_{l=1}^n|x_i^{(l)}-x_j^{(l)}|^2)^{\frac{1}{2}} \] 如果 \(p=1\),那么就是曼哈顿距离 \[ L_1(\boldsymbol{x}_i,\boldsymbol{x}_j)=\sum_{l=1}^n|x_i^{(l)}-x_j^{(l)}| \] 如果 \(p=\infin\),那么就是切比雪夫距离 \[ L_\infin(\boldsymbol{x}_i,\boldsymbol{x}_j)=\max|x_i^{(l)}-x_j^{(l)}| \] 但是考虑到样本各个特征属性之间的尺度并不一定,相关性也不一定,特别是各个维度之间的分布(方差)不同,此时就需要使用马氏距离。
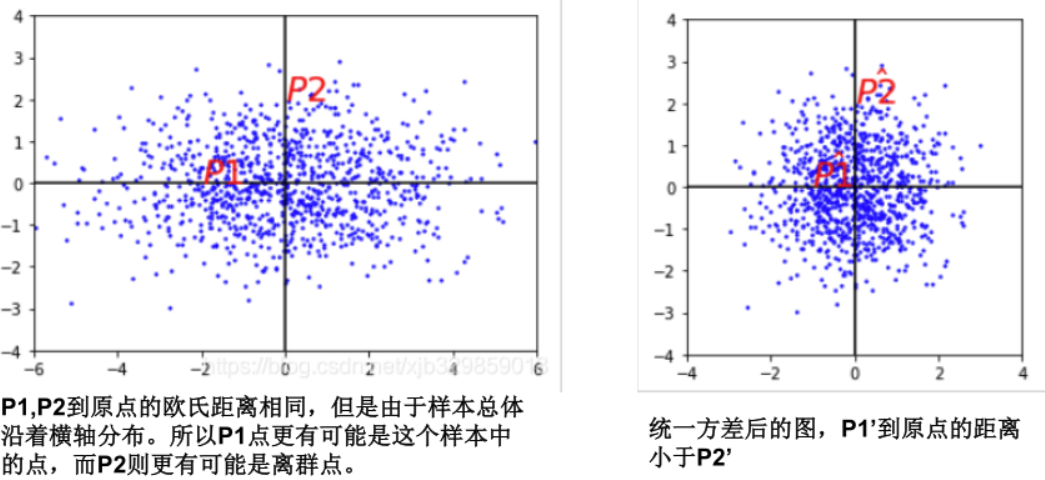
马氏距离公式为 \[ d_{Mahalanobis}=\sqrt{(\boldsymbol{x}-\boldsymbol{y})^T\Sigma^{-1}(\boldsymbol{x}-\boldsymbol{y})} \] 其中,\(\Sigma\) 是协方差矩阵。
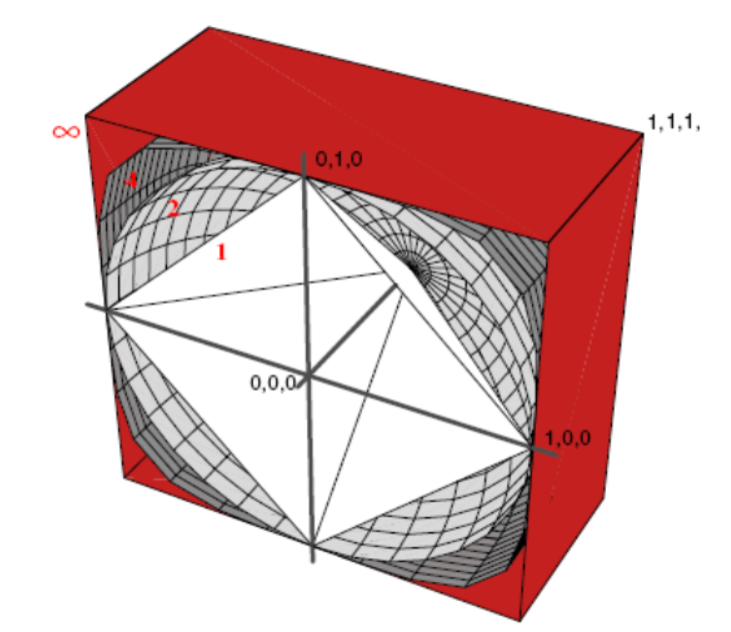
每一个彩色的平面由距离原点为 1 的点所形成。
代码
仅考虑两类样本,借助 numpy 计算闵可夫斯基距离。
1 | import numpy as np |
优缺点
如果 K 推向正无穷,那么就会利用全部的训练样本做训练,哪一类样本多就属于哪一类,会导致欠拟合,训练误差加大。
如果 K 值小,则模型复杂度较高,容易发生过拟合,而且预测结果对近邻实例点非常敏感,容易被噪声(noise)影响。
在应用中,通常采用交叉验证法来选取最优的 K 值,一般取奇数(保证不会出现两类邻居数量相等)。
KNN 不对样本做任何假设和分布估计,是一种懒惰算法,常用于作为一个基准分类器,比较容易解决多雷问题,且足够多训练数据下,KNN 可以实现任意分布数据的贝叶斯误差收敛(泛一致性)。
简言之,足够多训练数据下,KNN 分类器收敛到贝叶斯分类器(误差收敛)。
缺点是计算空间大,需要存储全部训练样本的信息,且计算开销大,需要计算测试样本与每一个训练样本的距离,同时同样容易陷入维数灾难,较少的训练样本性能一般也不太好。
主成分分析
简介
主成分分析(Principal Component Analysis, PCA)是统计学上常用的方法,通常是为了找出数据中最主要的元素和结构,去除噪音和冗余,将原有的复杂数据降维,揭示隐藏在复杂数据背后的简单结构。
PCA 的功能是简化复杂数据到低维空间,从而发现数据中隐藏的简单结构。
其简单且无参数限制,被誉为应用线性代数最价值的结果之一。
PCA 的目标是去除冗余,并发现重要特征。
不同于 LDA(最小化类内散度和最大化类间距离),它是单纯的降维。
方差
一般来说,方差是对于全体而言的,即 \[ \sigma^2=\frac{1}{n}\sum_{i=1}^n(x_i-\mu)^2 \] 其中,\(\mu\) 是全体均值,\(n\) 是全体数量。
但是考虑到,如果要计算所有的数据,负担太大,所以我们一般采用随机采样,然后计算这部分样本的方差,即样本方差 \[ s^2=\frac{1}{n-1}\sum^n_{i=1}(x_i-\mu)^2 \] 其中,\(n-1\) 是贝塞尔矫正,为了平衡自由度降低导致计算得到的样本方差减低。
但对于多维数据,我们并不能直接使用方差公式,此时需要改为用协方差 \[ Cov(\boldsymbol{x},\boldsymbol{y})=\frac{1}{n-1}\sum^n_{i=1}(x_i-\bar{x})(y_i-\bar{y}) \] 协方差矩阵用于描述多维度之间的相关性。
协方差绝对值越大,其相关关系越明显。
而方差的大小决定了数据在某一方向上的分散程度。
考虑一组数据
| 房价(百万元) | 面积(百平米) |
|---|---|
| 10 | 10 |
| 2 | 2 |
| 1 | 1 |
| 7 | 7 |
| 3 | 3 |
那么我们可以写出代码
1 | import numpy as np |
可以发现,两类样本之间相关程度大,且在 \(x\) 和 \(y\) 方向分散程度一致。
基变换
考虑原坐标系为直角坐标系,则其的基(basis)为 \[ \begin{bmatrix} 1&0\\ 0&1 \end{bmatrix} \] 考虑我们想将坐标 \((3,2)\) 转换到新正交基坐标系 \[ \begin{bmatrix} 1/\sqrt{2}&1/\sqrt{2}\\ -1/\sqrt{2}&1/\sqrt{2} \end{bmatrix} \] 那么仅需计算 \[ \begin{bmatrix} 1/\sqrt{2}&1/\sqrt{2}\\ -1/\sqrt{2}&1/\sqrt{2} \end{bmatrix}\begin{pmatrix} 3\\ 2 \end{pmatrix}=\begin{pmatrix} 5/\sqrt{2}\\ -1/\sqrt{2} \end{pmatrix} \] 同样的如果对三个点 \((1,1),(2,2),(3,3)\) 变换到新坐标系下,则计算 \[ \begin{bmatrix} 1/\sqrt{2}&1/\sqrt{2}\\ -1/\sqrt{2}&1/\sqrt{2} \end{bmatrix}\begin{pmatrix} 1&2&3\\ 1&2&3 \end{pmatrix}=\begin{pmatrix} 2/\sqrt{2}&4/\sqrt{2}&6/\sqrt{2}\\ 0&0&0 \end{pmatrix} \] 因此,基变换(坐标变换)可以被表示为矩阵相乘,使用矩阵对向量空间进行旋转(向量旋转)。
PCA 算法
基本数学工具是矩阵对角化。
考虑房价与面积的数据,我们对矩阵进行矩阵对角化,得到
1 | import numpy as np |
可以发现,矩阵对角化后显然仅有第一列数据是有意义的,而第二列数据的方差为 0,其是无意义的。
算法步骤如下:
- 将原始数据按列组成 \(n\times m\) 矩阵 \(X\)
- 将 \(X\) 的每一行进行去中心化(零均值化),即减去这一行的均值
- 求出协方差矩阵 \(C=\frac{1}{m}XX^T\)(除以 \(m-1\) 或者忽略都可以)
- 求出协方差矩阵的特征值和特征向量
- 将特征向量按特征值大小从上到下按行排列成矩阵,取前 \(k\) 行组成矩阵 \(P\),即将矩阵从 \(n\) 维降低到 \(k\) 维。
- \(Y=PX\) 即为降维到 \(k\) 维后的数据。
示例
考虑 5 个二维样本 \[ X=\begin{bmatrix} -1&-1&0&2&0\\ -2&0&0&1&1 \end{bmatrix} \] 去中心化(样本本身已经中心化了)。
求协方差矩阵 \[ C=\frac{1}{5}\begin{bmatrix} -1&-1&0&2&0\\ -2&0&0&1&1 \end{bmatrix}\begin{bmatrix} -1&-2\\ -1&0\\ 0&0\\ 2&1\\ 0&1 \end{bmatrix}=\begin{bmatrix} 1.2&0.8\\ 0.8&1.2 \end{bmatrix} \] 求特征值及特征向量 \[ \lambda_1=2,\lambda_2=0.4\\ c_1=(1,1)^T,c_2=(-1,1)^T \] 那么可以得到标准化正交基(标准化需要向量长度为 1)为 \[ P=\begin{bmatrix} 1/\sqrt{2}&1/\sqrt{2}\\ -1/\sqrt{2}&1/\sqrt{2} \end{bmatrix} \] 只选取特征值最大投影方向进行降维,那么 \[ Y=\begin{pmatrix} -3/\sqrt{2}&-1/\sqrt{2}&0&3/\sqrt{2}&-1/\sqrt{2} \end{pmatrix} \]
这里是计算 \(Y'=PX\),随后仅取第一行,因为第一行的特征值为 2,是最大值。
使用 numpy 可以写出代码如下
1 | import numpy as np |
聚类
原理
聚类目标:将数据集中的样本划分为若干个通常不相交的子集。
聚类既可以作为一个单独过程(用于找寻数据内在的分布结构),也可作为分类等其他学习任务的前驱过程。
K 均值算法
K 均值算法又称 K-means 算法,K 是聚类算法中类的个数,means 是均值算法,故算法本身是采用均值算法把数据分成 K 类的算法。
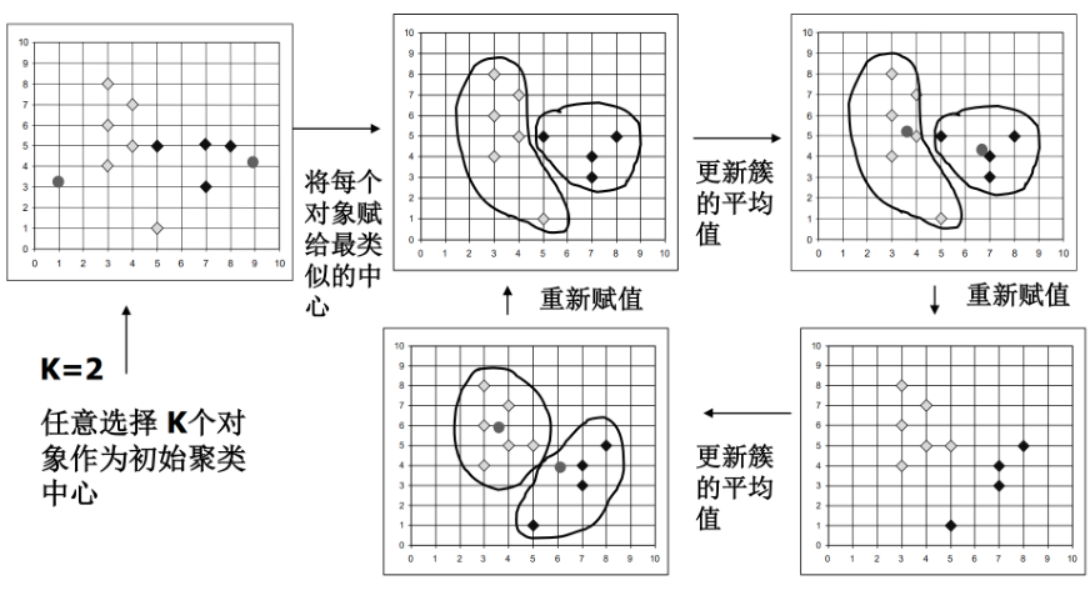
在聚类任务中,我们通常定义一个聚类准则函数来评价聚类结果的质量,可以类比线性分类中的损失函数。
如果聚类质量不满足要求,就要重复执行聚类过程,以优化结果。
最常用的是误差平方和准则 \[ J_c=\sum^c_{j=1}\sum^{n_j}_{k=1}\|x_k-m_j\|^2 \] 其中 \(m_j=\frac{1}{n_j}\sum^{n_j}_{j=1}x_j\),是该集合的中心,用来代表 K 个类型。
而 \(J_c\) 是样本和集合中心的函数,在样本集 \(X\) 给定的情况下,\(J_c\) 的取值取决于 \(c\) 个集合中心。
试验样本聚合成 \(c\) 个类型时,所产生的总误差平方和 \(J_c\) 越小越好。
考虑以下西瓜数据集,来运行 K 均值算法。
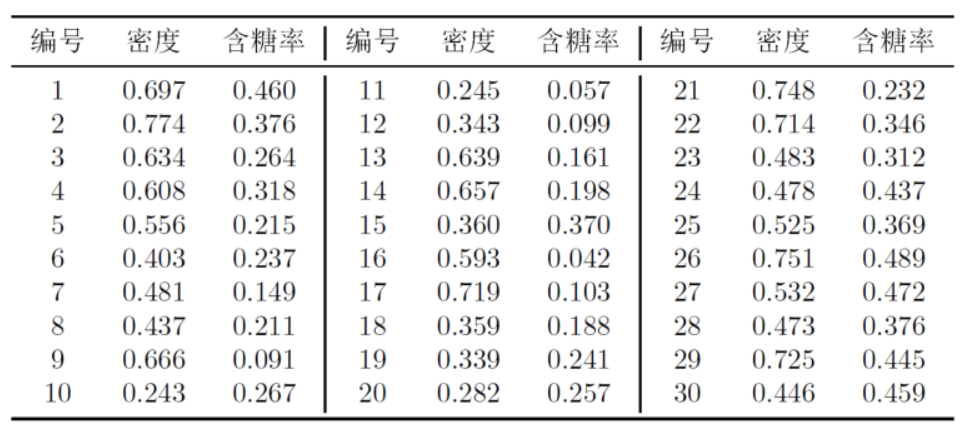
1 | import numpy as np |